2022年6月17日 国立大学法人東京大学大気海洋研究所 プレスリリースにて
地球表層圏変動研究センター 吉澤晋准教授らが発表を行い、記事が掲載されました。
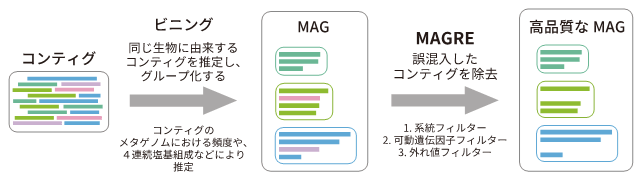
図1.MAGを解読する手法の概略と、MAGREによるゲノム品質の向上を図示した。3つのフィルターを備えたMAGREにより、誤ってMAGに混入したコンティグを検出・除去することが可能である。
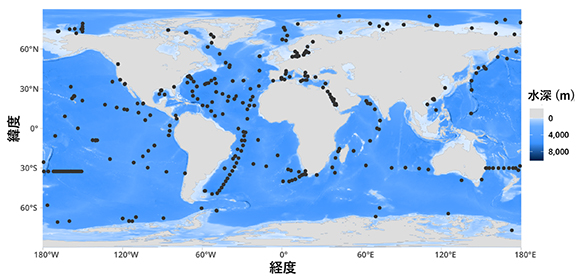
図2.世界の各海域にまたがる、2,057メタゲノムのサンプリング地点を黒点で示した。
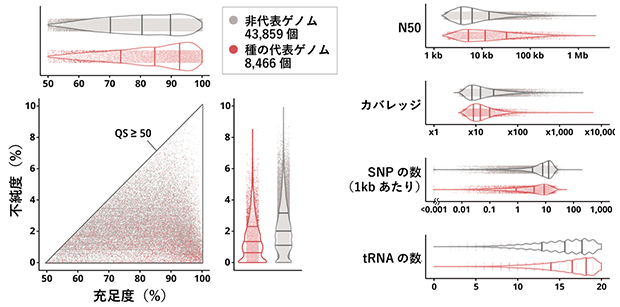
図3.OceanDNA MAGを、種の代表ゲノムと非代表ゲノムに分けて、それぞれのゲノムの特徴となる数値を示した。充足度(completeness)と不純度(contamination)はCheckMというソフトウェアを用いて計測した。充足度の中央値は80%以上、不順度の中央値は2%未満となっており、OceanDNA MAGカタログに含まれるゲノムの品質は非常に高い。また、カバレッジやSNPの数は、元のメタゲノム配列をゲノムに貼り付けることにより算出した。
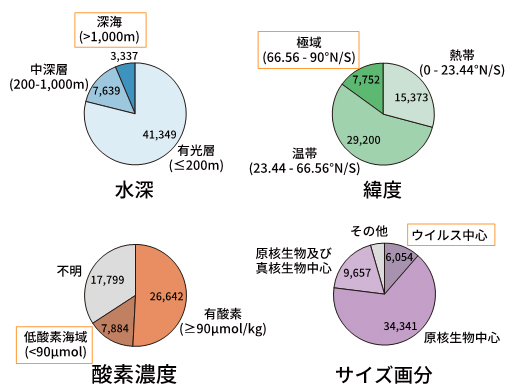
図4.OceanDNA MAGの由来となる海洋環境を、水深、緯度、酸素濃度、サイズ画分別で区分化したもの。深海(>1,000m)、極域、低酸素海域、ウイルス中心(0.2μmを透過したサンプル)など、これまでにゲノム情報があまり得られていない海洋環境・サンプルからも多くのゲノムを解読した。
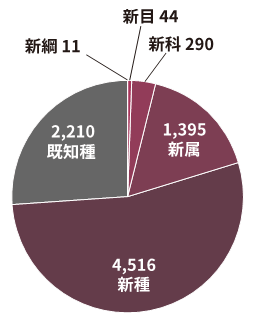
図5.得られた8,466種を、新規性によって分類した際の内訳を示した。新綱相当が11種、新目相当が44種、新科相当が290種と、多数の種が非常に高い新規性を示した。また、全体の73.9%が新種に相当する。
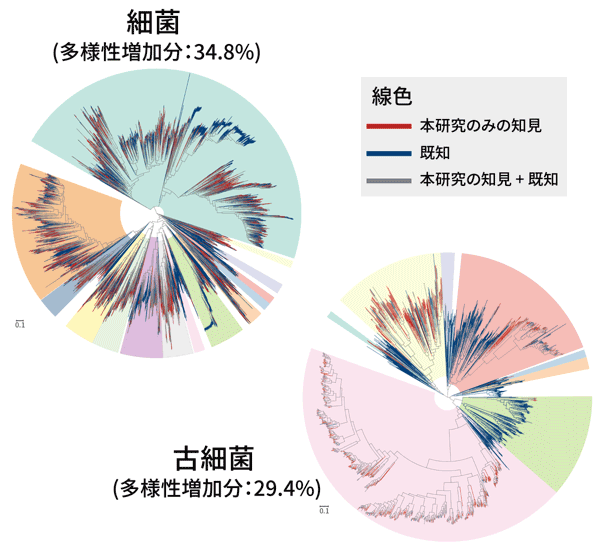
図6.OceanDNA MAGカタログによるゲノム多様性の増加分について、系統樹を用いて可視化した(左:細菌、右:古細菌)。赤線部分は、OceanDNA MAGカタログで新しく発見されたゲノム多様性であることを示す。多様性増加分は、既知の多様性(青線+灰色線)に対する発見された多様性(赤線部分)の割合を示す。図中で扇形は、色別に門(phylum)レベルの系統を示す。図の明瞭さを保つため、種の代表ゲノムのみを用いて可視化した(細菌:14,764ゲノム、古細菌:1,377ゲノム)。
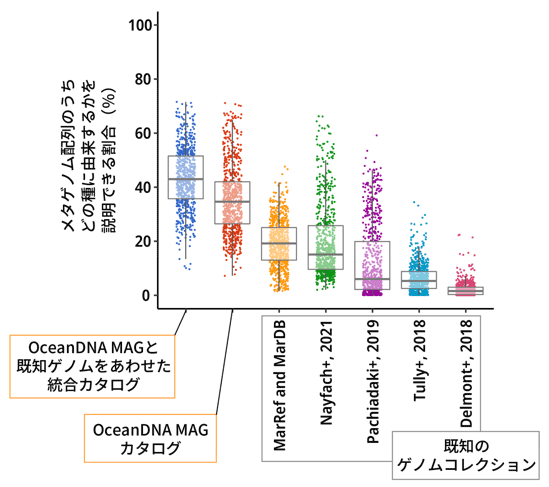
図7.各ゲノムコレクションを用いて、各メタゲノム配列が、どの原核生物に由来するかを説明できる割合を図に示した。図中の点は、各メタゲノム(原核生物中心となる画分に由来する732メタゲノム)に関する割合を示す。OceanDNA MAGカタログでは、26.5〜42.0%(四分位範囲)となり、どの既知のゲノムコレクションよりも高い割合となっている。また、OceanDNA MAGカタログと既知のゲノムコレクションを合わせた統合カタログでは、34.2〜49.6%(四分位範囲)となった。
掲載内容
「微生物ダークマター」を解き明かす ―世界最大の海洋微生物ゲノムカタログ―
発表のポイント
♦膨大なメタゲノムデータ(注1)から高い品質で個々の微生物のゲノムを解読する手法を新たに開発しました。
♦開発した手法を大規模な海洋メタゲノムデータ(約29兆塩基対)に適用した結果、59門(注2)にまたがる5万以上の原核生物ゲノム(注3)の解読に成功し、解読されたゲノムには新綱(注4)相当のものを含む、高い新規性が見出されました。
♦得られたゲノム情報を、世界最大の海洋微生物ゲノムカタログ「OceanDNA MAGカタログ」として公開しました。
♦本研究は、「微生物ダークマター」(注5)に相当するゲノムを多数解読することで、海洋微生物の知られざる多様性を解明しました。OceanDNA MAGカタログは、海洋生態系を理解し、さらには地球環境変動の要因解明や予測のための重要な情報基盤となります。
発表概要
海洋環境には「微生物ダークマター」と呼称される、未知の微生物が数多く残されています。これらの微生物がどのような生態を持ち、どういった進化を遂げてきたのかは、謎に包まれてきました。
東京大学大気海洋研究所の西村陽介特任研究員(研究当時)と吉澤晋准教授は、メタゲノムデータを用いて、個々の微生物ゲノム(一般に、MAG(注6)と呼ばれる)を解読する手法を開発しました。この手法を用いて、公共データベースから入手可能な、様々な海洋環境に由来する大規模メタゲノムデータ(約29兆塩基対)を解析しました。その結果、59門にまたがる5万以上の原核生物ゲノムの解読に成功し、得られたゲノムの情報を整備して、約15,000ゲノムを収載する海洋微生物ゲノムデータベースMarDBを大幅に上回る、世界最大の海洋生物ゲノムカタログ「OceanDNA MAGカタログ」として公開しました。
本研究は、「微生物ダークマター」に相当するゲノム配列を多数解読することで、海洋原核生物ゲノムの多様性に関する知識を大幅に拡充しました。OceanDNA MAGカタログは、海洋微生物の生態や進化の過程の解明に貢献するとともに、地球環境保全に取り組むための重要な知識基盤となることが期待されます。
発表内容
【研究の背景】
生命誕生以来、微生物の活動は地球環境の形成に大きな影響を与えてきました。例えば、微生物は地球規模の物質循環において重要な役割を果たしていることが知られています。近年では、地球環境保全への関心の高まりのなかで、環境変動に対して微生物がどのような「応答」を示すかに注目が集まっています。特に、海洋は地球の表面積の約70%を占め、10の30乗と推定されるおびただしい数の微生物が生息しています。海洋微生物の多様性や、その生態を理解することは、今後の地球環境を保全・維持していくために必要不可欠な課題です。しかしながら、海洋環境には「微生物ダークマター」と呼称される、正体不明な微生物が多く残されており、海洋微生物の全体像は謎に包まれています。
メタゲノムデータは、環境サンプルから抽出したDNAを無作為に解読することで得られる配列データであり、微生物ダークマターのゲノムを解読するために必要な情報が含まれています。近年、配列解析の技術革新とコスト低下によって、大規模なメタゲノムデータが公共データベース上に蓄積されていますが、配列情報を「読み解く」ことは容易ではありません。データを読み解くための方法論として、メタゲノムデータから個々の微生物ゲノム(MAG)を解読する手法が開発されており、先行研究によって、海洋メタゲノムから数千個のMAGが得られています。しかしながら、MAGに別の生物由来のコンティグ(注6参照)が混在していることがあり、MAGの品質向上が技術的な課題となっています。
【研究内容】
東京大学大気海洋研究所の西村陽介特任研究員(研究当時)と吉澤晋准教授は、東京大学未来社会協創(FSI:Future Society Initiative)プロジェクト『オーシャンDNA』の一環として、メタゲノムデータからMAGを解読する手法を新たに開発しました。本手法ではMAGの品質を向上させるための様々な工夫がなされています。特に、本研究で開発したMAGRE(図1)は、コンティグの様々な特性を解析することで、ゲノムに誤って含まれたコンティグを検出・除去するためのソフトウェアです。この技術開発により、高品質なMAGをより多く解読することが可能となりました。MAGREのソースコードは、GitHub(注7)にて一般公開されています。
本手法によって、世界の各海域で行われた研究プロジェクトに由来する2,057の海洋メタゲノム(図2)に由来する配列データ(約29兆塩基対)の解析を行いました。その結果、原核生物59門にまたがる52,325個の高品質なゲノムの解読に成功しました(図3)。本研究では多様な海洋環境に由来するメタゲノムデータを使用しており、これまでにゲノム情報があまり得られていない海洋環境から多くのゲノムを解読することができました。例えば水深1,000m以深の深海から3,337ゲノム、極域から7,752ゲノム、低酸素海域から7,884ゲノムが解読されています(図4)。得られたゲノムは、その類似性を評価することによって、8,466生物種に由来することがわかりました。この8,466種について、既知のゲノムと比較して新規性を評価すると、73.9%が新種相当であり、新綱相当が11種、新目相当が44種含まれるなど、非常に高い新規性が見出されました(図5)。得られたゲノム情報を整備し、OceanDNA MAGカタログとして公開しました。
得られたゲノムの多様性を評価したところ、OceanDNA MAGカタログによって海洋原核生物ゲノムの多様性に関する知識が34.2%も拡大したことがわかりました(図6)。また、OceanDNA MAGカタログは、海洋メタゲノム配列の26.5 〜 42.0%(四分位範囲)について、その由来となる生物種を説明することができました(図7)。これは過去に発表された他の海洋原核生物ゲノムデータセットよりも高い割合となっています。
【社会的意義・今後の予定】
本研究で開発したゲノム品質向上のためのツールMAGREは汎用性が高く、海洋のみならず様々な環境から得られたゲノムの情報解析において利用されることが期待されます。また、OceanDNA MAGカタログは、海洋微生物の生態や進化の軌跡の解明に貢献するだけでなく、地球環境保全に取り組む上で重要な知識基盤となることが期待されます。実際に、発表者らは本研究で得られたゲノム情報を用いて、海洋の光合成細菌の全容解明に向けた解析などの関連研究を広く展開しており、本ゲノムカタログを活用した一層の研究成果が期待されます。
発表雑誌
雑誌名:Scientific Data
論文タイトル:
The OceanDNA MAG catalog contains over 50,000 prokaryotic genomes originated from various marine environments
著者:
Yosuke Nishimura*, Susumu Yoshizawa
doi: https://doi.org/10.1038/s41597-022-01392-5
用語解説
・注1 メタゲノムデータ:
環境サンプルから抽出したDNAをシークエンサーで解読することで、環境中の微生物を解析することをメタゲノム解析と呼ぶ。解読された配列データはメタゲノムデータと呼ばれ、その多くが公共データベース上で公開される。
・注2 門:
門(phylum)は生物の分類における大きな単位。例えば昆虫や甲殻類は同じ節足動物門、ヒトやは虫類は同じ脊索動物門に属する。本研究が準拠しているGTDB (Genome Taxonomy DataBase) release 95の系統分類においては、細菌は131門、古細菌は18門から構成される。
・注3 原核生物:
細菌と古細菌を合わせた分類群。生物は大きく分けて、原核生物と真核生物に二分される。また、ウイルスも遺伝物質(つまりDNAなどの核酸)を持つため、海洋メタゲノムから得られる配列は、この3者のいずれかに由来すると考えられる。
・注4 新綱:
綱(class)は原核生物の分類において、門の次に大きい分類単位。例えば上記のヒトとは虫類は別の綱に属する。ここで言う新綱とは、現在の綱の分類にいずれも当てはまらないほどにゲノムの新規性が高いことを意味する。
・注5 微生物ダークマター:
生物系統不明の、未知な微生物であり、宇宙に存在する未知の暗黒物質(ダークマター)に例えられる。海洋には、多くの微生物ダークマターが存在すると考えられる。
・注6 MAG:
Metagenome-Assembled Genomeの略。メタゲノム解析では、得られたリード配列(断片化された塩基配列であり、配列解読の単位)をつなぎ合わせて(アセンブルして)「コンティグ」と呼ばれるゲノム断片配列を得ることができる。そのコンティグのうち、同じ生物に由来すると推定されるものをグループ化する(ビニングと呼ばれる)ことで、一つのゲノムとして解釈されたものをMAGと呼ぶ(図1参照)。
・注7 GitHub:
様々な分野で利用されるソフトウェア開発のプラットフォームであり、ソフトウェアの管理や、コードの公開ができるwebサービス。MAGREは以下のURLにて公開されている。https://github.com/yosuken/MAGRE
添付資料
関連リンク
・大気海洋研究所 プレスリリース(2022年6月17日)









